елементи
абстрактно
Материали и методи
Етично изявление
Цялата работа с животни, описана в това проучване, е одобрена от Институционалния комитет по грижата и използването на животните (IACUC) към Университета в Арканзас и всички експерименти са извършени в съответствие с одобрените насоки и разпоредби.
Проектиране на експеримент с животни
Вземане на кръв и екстракция на ДНК
Взети са кръвни проби от 5 привидно здрави птици на кошара на 14, 41 и 49 ден за анализ на микроорганизми, за да се получат 240 кръвни проби (5 птици на кошара × 16 писалки на възраст за вземане на проби x 3 възрастови категории). Още 12 кръвни проби бяха взети от птици, подложени на BCO (n = 12; 5 и 7 проби на възраст от 41 и 49 дни, съответно), което позволява да бъдат анализирани общо 252 проби за профилиране на 16S рРНК. Кръвни проби бяха взети асептично от вената на крилото с помощта на EDTA Vacutainers. Един ml кръвна проба се центрофугира (5000 rpm в продължение на 5 минути при стайна температура) с помощта на микроцентрифуга и 200 μl buffy слой се събира в стерилна ламинарна камера и се съхранява при -20 ° C. Геномна ДНК се екстрахира от buffy coats използвайки BiOstic® Bacteremia DNA Isolation Kit (MoBio) в съответствие с инструкциите на производителя. ДНК пробите се анализират с помощта на Qubit 2.0 флуорометър (Life Technologies) за количество и чистота и се съхраняват при -20 ° C.
PCR протокол за амплификация на 16S рРНК ген
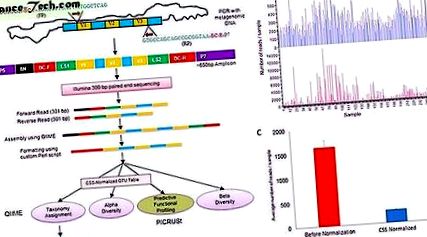
( а ) Блок-схема, представяща подготовката на библиотеката, форматиране на показанията, приемливи за QIIME анализ, които бяха допълнително анализирани с помощта на LefSe и PICRUSt. * За анализ PICRUST е използвана стандартизирана таблица на OTU. (FP = праймер, RP = обратен праймер, V = променлива област на 16s РНК гена, P5 и P7 = праймери на последователността Illumina, N = случаен нуклеотид, BC-F = баркод на праймера, BC-R = обратен праймер баркод) и LS = свързваща последователност). ( б ) Стълбовидна диаграма, показваща разпределението на 252 проби преди (червено) и след нормализиране (синьо) на таблицата OTU. ° С ) Стълбовидна диаграма, показваща средния брой отчитания на проба със стандартна грешка преди (червено) и след нормализиране (синьо).
Изображение в пълен размер
Анализ на данни
Блок-схема, описваща анализа на 16S рРНК последователността е показана на ФИГ. 1а. Четенето напред и назад на сдвоения край от последователността на Illumin беше съставено, използвайки количествени прозрения в микробната екология, QIIME 1.9.1 25, чрез скрипта join_paired_ends.py, използвайки метода fastq-join. След това последователностите от баркодове за четене напред (BC-F) и обратно (BC-R) бяха присъединени и присъединени (5 'край) към сглобената последователност (3' край) след отстраняване на 8 нуклеотидни случайни последователности и 27F и 533R последователности от праймери от собствения скрипт на Perl, който създаде fastq файл, съвместим с последващ анализ с помощта на QIIME.
Четенията бяха групирани с помощта на UCLUST 26 и оперативните таксономични единици (OTU) с възможност за затваряне на референцията бяха извършени с помощта на изданието 13_8 на базата данни GreenGenes. Таблицата OTU BIOM (матрица за биологично наблюдение) беше нормализирана с помощта на QIIME (normalize_table.py), използвайки метода на кумулативната сума (CSS), който след това беше използван за таксономични назначения, оценка на алфа разнообразието, идентификация на биомаркери и прогнозиране на съдържанието на функционален метагеном. Индексите на алфа разнообразието бяха сравнени с помощта на еднопосочен дисперсионен анализ (ANOVA), а post hoc анализът беше извършен по метода на Tukey-Kramer HSD. Анализът на сходството (ANOSIM) между групите метаданни беше извършен, като се използва непретеглената метрика за разстояние UniFrac с QIIME (compare_categories.py). В допълнение бяха извършени главни координационни анализи (PCoA), за да се оцени бета разнообразието с QIIME (24), като се използват претеглени и непретеглени UniFrac метрики при различни дълбочини на вземане на проби.
Идентификацията на биомаркер е извършена, като се използва величината на ефекта на линеен дискриминант анализ (LEfSe) 27. Метагенното функционално съдържание на микробиомите на пилешката кръв беше предсказано с PICRUSt (филогенетично изследване на общности чрез реконструкция на ненаблюдавани състояния), използвайки 16S rRNA генни последователности 28. Бактериалната мрежа е визуализирана с помощта на Cytoscape 3.2.1.
резултатът
Демултиплексирането и качественото филтриране на QIIME произвеждат 4, 153, 965 сглобени последователности, вариращи от 40 до 580 bp със средна дължина 469 bp, което показва наличието на химерни последователности поради отклонено PCR усилване или несъответствие. В това проучване използвахме затворения референтен метод на OTU, тъй като изследваме нов тип микробиоми, за които структурата на общността, разнообразието и членството са до голяма степен неизвестни. Въпреки това, ние също сравнихме резултатите, получени при използване на затворен референтен метод с отворените референтни методи и методите за ново бране на OTU (допълнителна таблица 2). Въпреки че отворената справка и методът за ново избиране на OTU доведоха до по-голям брой OTU (съответно средно 196 и 218), както и до по-високи стойности на извадката, бяха регистрирани значителни пропорции (средно 72, 5 и 79, съответно%). ), които не бяха включени в нито една таксономична група в настоящата база данни GreenGenes 13_8. В допълнение към ефективното премахване на всички шумови или химерни последователности в последващия анализ, ние предпочетохме затворена референтна OTU колекция пред другите два метода.
Затворената референтна таблица на OTU имаше средна дълбочина на пробата от 1,621 за всяка проба (± 139, 07), както е показано на фиг. Таблицата OTU беше нормализирана с помощта на кумулативно мащабиране (CSS) с QIIME (Фиг. 1b, c). Въпреки че средните стойности на пробите са намалени чрез нормализиране повече от 6 пъти (266, 55 на проба), те също така намаляват стандартната грешка (± 7, 01). CSS нормализираната OTU таблица беше използвана за присвояване на таксономия, оценки на алфа разнообразието, LEfSe анализ и PICRUt анализ. Анализът на бета разнообразието беше извършен при различни дълбочини на вземане на проби и ANOSIM с 999 пермутации, използвайки непретеглена метрика UniFrac при същата дълбочина на вземане на проби от 400 кадъра на проба.
Присвояване на таксономия
а ) Стълбовидна диаграма на нивото на багажника с диаметър. Различните букви над всяка колона показват значителни разлики (ANOVA, тест на Туки-Крамер, стр. 4 .
Кутия ограда експериментална категория с дистанционно. Полетата показват средния, 25-ия и 75-ия персентил със стойности извън долната и горната крайност. Кутиите с различни букви са значително различни (стр
а ) PCoA графика на здрави (n = 171) срещу BCO (n = 9) пилета, използващи претеглени UniFrac показатели при една и съща дълбочина на вземане на проби от 400 прочетени за проба. ( б ) PCoA графика от 14 дни (n = 30), 41 дни (n = 41) срещу 49 дни (n = 36) стари пилета с претеглени UniFrac метрики при същата дълбочина на вземане на проби от 1 000 кадъра на проба. ( ° С ) PCoA диаграма на стартер (n = 67) срещу пилета за финишър (n = 154), хранени с диети, използващи непретеглена метрика UniFrac при същата дълбочина на вземане на проби от 200 кадъра на проба.
Изображение в пълен размер
Значителна разлика в бактериалните съобщества на здрави и BCO пилета също се илюстрира чрез йерархично групиране. BCO пилета с 49-дневни пилета бяха разделени от здрави пилета. Въпреки това, 41-дневните BCO пилета не показват характер на значително групиране (Фиг. 5а). Мрежовият анализ между пилета (252 проби) и OTU (бактериални видове) показва известна степен на разделителна способност в моделите на взаимодействие между BCO и здрави пилета (фиг. 5б), което предполага различни бактериални съобщества.
а ) Йерархично групиране на здрави и BCO пилета въз основа на микробна кръв. Филогенетичното дърво е създадено с помощта на софтуера FigTree V1.3.1 с претеглени UniFrac метрики, използвайки данни от предварително нормализиране. Зелените и червените линии представляват BCO пилета за 41 и 49 дни. Черните линии са здрави пилета. Числото в края на реда показва идентификационния номер на пробата на пилето. б ) Бактериална мрежа от микроорганизми от пилешка кръв, произведени с Cytoscape V3.2.1. Червените, сините и белите възли представляват BCO пилета, здрави пилета и OTU. Зелената граница е BCO пилешката мрежа, а оранжевият ръб е при здрави пилета.
Изображение в пълен размер
Отбелязахме, че всички BCO птици са само от две възрастови групи (ден 41 и 49) и две групи (W35-56 и W1-56). Поради това извършихме допълнителен анализ на бета разнообразието, като използвахме подгрупа от 77 проби, принадлежащи към тези възрастови групи и към групи, базирани на устни, включително 65 здрави птици и 12 BCO птици. ANOSIM анализ, базиран на UniFrac претеглени показатели, показва по-голямо разделяне между BCO и здрави пилета (R = 0,5293, p = 0,001) в сравнение с общия набор от данни (n = 252). Графиката PCoA, показана на фиг. 6 също подкрепя резултата от ANOSIM анализа.
PCoA графика на здрави (n = 51) срещу BCO (n = 9) пилета, използващи претеглени UniFrac метрики при същата дълбочина на вземане на проби от 400 прочетени за проба.
Изображение в пълен размер
BCO биомаркери
Таксономични групи, които се различават в изобилие между здрави и BCO пилета, бяха идентифицирани, като се използва величината на ефекта на линейния дискриминант (LEfSe) с a = 0,05, оценка LDA от поне 2 и относително число над 0,1. Общо 26 функции имаха значително различни количества между здрави и BCO пилета. На ниво род, микробиодите на BCO в кръвта на пилета са обогатени диференциално със Staphylococcus, Granulicatella и Microbacterium, докато здравите пилета са обогатени с Pseudomonas, Enhydrobacter и Aquabacterium (Фиг. 7b). Установихме също, че щамът Firmicutes е обогатен с пилета BCO. По същия начин Alphaproteobacteria беше силно обогатена при BCO пилета, докато Betaproteobacteria и Gammaproteobacteria при здрави пилета на ниво клас (Фиг. 7b).
а ) Таксономична кладограма, направена от анализ на LEfSe. Червеното и зеленото съдържат таксони, обогатени с BCO и здрави пилета. Яркостта е пропорционална на количеството таксон. ( б ) Данъците, обогатени с BCO пилета, са показани в червено с отрицателен LDA резултат и здрави пилета в зелено състояние с положителен LDA резултат (> 3, 5 и в двата случая). Таксонът между двете подчертавания се предлага като име на базата данни GreenGene.
Изображение в пълен размер
Предполагаем функционален генетичен капацитет на микроорганизмите от пилешка кръв
( а ) Относителен брой нива на KEGG COG на ниво 1 в микробиомите на пилешката кръв. ( б ) Относително изобилие (≥ 0, 5) на пътя KEGG на ниво 3 на функционалните пътища, създадени от софтуера JMP. ( ° С ) диференцирани знаци (категория KEGG COG, относително изобилие ≥ 0, 5), получени с използване на LEfSe с оценка LDA ≥ 2, 8.
Изображение в пълен размер
дискусия
Това е първото изчерпателно проучване за анализ на бактериалните микроби, които съществуват в кръвта на гръбначните животни, различни от човека. През последните години нараства интересът към характеризиране на микробиона, свързан с различни тъкани на тялото при различни здравословни условия. Части от тялото, които се считат за стерилни, като кръв 30, стомах 31, пикочен мехур 32, бели дробове 33, кости, стави 4 и гърди 34, имат своя оригинален микроорганизъм. Дисбиозата на тъканната микробиоза е свързана с различни заболявания, включително сърдечно-съдови заболявания 10, диабет 35, безалкохолно чернодробно заболяване на черния дроб (NASHD) 36, възпалително заболяване на червата (IBD) 37, псориазис 38, затлъстяване 39, детска астма 39, функционално заболяване на червата 40 и колоректален рак 41. Тук изследвахме микробиозата на пилешка кръв, за да идентифицираме потенциални бактериални биомаркери, свързани с BCO.
Резултатът от анализа на данните в това проучване показва съществуването на бактериални съобщества, състоящи се от 30 до 40 OTU в кръвта на пилета-бройлери, независимо от възрастта и други условия на околната среда или гостоприемника. Анализ на бета разнообразието (фиг. 4а и 6), йерархичен клъстер анализ (фиг. 5а) и анализ на бактериалната мрежа (фиг. 5б) на базата на микробна кръв и йерархично клъстериране на базата на предсказан метагеном на микробион на кръв (фиг. 8б) предполагат, че бактериалните съобщества в кръвта на BCO птиците се различават от общностите при здрави птици, което показва наличието на определени селективни налягания, които допринасят за изместването на кръвните микробиомери в BCO птиците. Обаче само цялото PD дърво показва значителна разлика в алфа разнообразието между BCO и здравите птици, което е в съответствие с филогенетично отдалечената микробиоза на кръвта при BCO птици, както е показано в анализа на бета разнообразието (Фигури 4а и 6).
В нашето непубликувано придружаващо проучване, кръвни проби от същото стадо бяха директно инокулирани върху богата агарова среда. Резултатът показа, че броят на колониите на 49-ия ден е постоянно по-висок на телени подове (L35W и W56) в сравнение с постелки (L56), което предполага, че напрежението върху теления под насърчава бактериемията вероятно чрез бактериална транслокация през чревния епител. Освен това броят на колониите на 49-ия ден също е бил постоянно по-висок при куците птици в сравнение със здравите птици. Тези резултати показват силна корелация между напрежението на пода от тел, тежестта на бактериемията и куцотата на BCO.
Едно практическо приложение на това проучване е да се идентифицират бактериални биомаркери, които могат да се използват за идентифициране на отделни пилета-бройлери в по-ранна възраст, които са податливи на развитието на BCO в по-късна възраст. Всъщност, анализът на данните в това проучване идентифицира таксономични групи на различни нива, които са значително обогатени с BCO птици в сравнение със здрави птици. Интересното е, че родът Staphylococcus е една от 18 черти (включително 3 рода), значително обогатени в проби от BCO, подчертавайки значението на този род, който често е изолиран от BCO лезии 3, 4, 5. Наскоро Al-Rubaye et al. 44 заяви, че предизвикателството на бройлери с S. agnetis, който е най-често изолираният стафилокозен вид от BCO лезии в своето проучване, значително е увеличило нараняването от 10 (Control) на 40%, докато предизвикателството с друг изолат на Enterococcus faecalis е намалило честота на мускулно-скелетни нарушения, Това може да показва потенциалното значение на S. agnetis като патоген на BCO при пилета-бройлери, въпреки че данните за профилиране на ген 16S rRNA не предоставят значима информация за видовете Staphylococcus поради ограничена таксономична резолюция.
В това проучване обаче BCO е открит само при птици на възраст 41 и 49 дни и следователно бактериалните биомаркери имат ограничена стойност за ранната диагностика на чувствителни към BCO птици. Този аспект трябва да бъде внимателно разгледан в експериментален план за бъдещи проучвания, за да се позволи идентифицирането на потенциални бактериални биомаркери в проби от кръв на млади птици, които предсказват развитието на BCO в по-старите възрастови групи.
Анализираните в това проучване кръвни микроби имат значителен ефект върху здравословното състояние на пилетата-бройлери, включително патогенезата на BCO, както е показано в това проучване, както и други заболявания или стрес при пилета-бройлери.
Повече информация
Как да цитирам тази статия: Mandal, RK et al. Изследване на микробна кръв и нейната потенциална връзка с бактериална хондронекроза с остеомиелит (BCO) при бройлери. Sci. Представител. 6, 25882; doi: 10.1038/srep25882 (2016).