елементи
абстрактно
През последните 30 години извършихме много основни проучвания върху две Oryza sativa subsp. indica, Zhenshan 97 (ZS97) и Minghui 63 (MH63). За да подобрим разделителната способност на много от тези проучвания, създадохме две референтни референтни геномни групи, използвайки най-съвременните технологии за секвениране. Използвайки технологията PacBio SMRT, ние създадохме повече от 108 (ZS97) и 174 (MH63) Gb сурови последователни данни от 166 (ZS97) и 209 (MH63) групи BAC клонинги и генерирахме
74 (MH63) Gb данни за последователност на целия геном, сдвоени в крайна последователност (WGS) с технология за секвениране Illumina. С тези данни ние успешно съставихме два стандартни платинени стандарта, които бяха публикувани. Ето пълните набори от сурови данни, използвани за генериране на тези две референтни геномни групи. Тези набори от данни могат да се използват за тестване на нови програми за по-добро сглобяване и анотиране на генома, за да помогнат за откриването на нови перспективи за структурата, функцията и еволюцията на генома и за осигуряване на необходимата подкрепа за биологични изследвания като цяло.
Обобщение на метаданните
Изтеглете файла с метаданни
Достъпен за машината файл с метаданни, описващ отчетени данни (формат на ISA карта)
Предистория и резюме
методи
Изграждане на BAC библиотека и последователно секвениране
Двете BAC библиотеки, използвани в това проучване, бяха публикувани преди това 11. Накратко, частично усвоената (т.е. Hin dIII) и избраната по размер геномна ДНК от всеки сорт се клонира в Hin dIII мястото на pAGIBAC1 и се трансформира в компетентни Escherichia coli DH10B T1 клетки. И двете библиотеки, наречени OSIZBa (ZS97) и OSIABa (MH63), съдържаха 36 864 BAC клонинги, средните размери на вложки бяха
125 kb (MH63) и бяха покрити
10, 7 × (MH63) от всеки геном 11. Освен това за първата половина на всяка библиотека 11 бяха генерирани 33, 969 (ZS97) и 35, 549 (MH63) двупосочни BAC крайни последователности (BES).
Физически карти
Описаните по-горе физически карти с ниско покритие (PM), използващи метода на пръстови отпечатъци SNaPshot, са описани по-горе11. Преобразувахме двете PM, използвайки метода за профилиране на целия геном KeyGene (WGP) 12. WGP FingerPrint Contig (FPC) PMs са конструирани в четири стъпки: (1) Подготовка на BAC ДНК, (2) подготовка на WGP BAC плазмиди с адаптери за индексиране и секвениране, (3) Illumina секвениране и (4) обработка на биоинформатика. В стъпка 4, използвайки скриптове за декодиране на WGP, бяха деконволюирани 99 996 (ZS97) и 103 609 (MH63) уникални WGP тагове и бяха маркирани 32 829 (89,1%) и 30 749 (89,3%) BAC в библиотеките ZS97 и MH63, респ. Използвайки маркери за WGP последователност за всеки BAC клонинг от всяка библиотека, бяха създадени две нови PM с FPC софтуер 13 (версия 9.4). След ръчно редактиране и интегриране с предишни SNaPshot PM, подобрените ZS97 и MH63 PM се състоят от 539 и 401 контига, съдържащи 28, 372 и 24, 519 клонинги и 4 457 и 6 230 клонинги като единични. Общите размери на контига са оценени на 342 Mb за ZS97 (N50 = 940 kb) и 349 Mb за MH63 (N50 = 1,270 kb).
Последователност на клонирането на PacBio BAC
Минималните BAC клонове на път за павиране (MTP) от всеки PM бяха избрани автоматично с помощта на персонализиран скрипт и ръчно подредени в ястия на библиотека на MTP с етикет OSIZBzz (ZS97) и OSIABzz (MH63) и съхранявани при -80 ° C. 4.751 BAC MTP клонинги бяха събрани за ZS97 и MH63. Пълните списъци с MTP клонинги са налични в Допълнителна таблица 1a-b.
За да се секвенира PacBio BAC клонинг, MTP BAC клонингите се инокулират в 96-гнездови блокове за растеж с дълбоки ямки, отглеждат се за една нощ при 37 ° С, центрофугират се за гранулиране на клетките и след това се съхраняват при -80 ° С до употреба. След това BACs бяха формирани чрез сливане на кладенци от замразени блокове в една от шест конфигурации: т.е. асоциации на единични кораби (12 BACs на пул), две асоциации на редове (24 BACs на пул), четири групи колони (32 BACs на пул), 6 групи колони (48 BAC на пул), осем асоциации на колони (64 BAC на пул) или групи на пълен пансион (96 BAC на пул). След това ДНК се екстрахира от всяка смес, като се използва стандартен протокол за изолиране на алкален лизис плазмид плазмид ДНК 14. Бяха секвенирани общо 166 (ZS97) и 209 (MH63) пула (вижте нашата подробна схема за обединяване в Допълнителна таблица 2a-b). Използвайки 16 μg обединена плазмидна ДНК, библиотеките на PacBio последователности бяха приготвени съгласно следните протоколи на производителя, както е описано за 20 kb Blue Pippin матричен препарат. SMRT секвенирането беше извършено на PacBio RSII инструмент, използвайки P5/C3 химия на последователността и 3 часа филми.
Производство на сурово четене с PacBio
Анализи на подвериги за двете групи на ZS97 и MH63 BAC последователности бяха извършени с помощта на PacBio SMRT Portal (версия 2.3.0). За ZS97 данните от 227 SMRT клетки (които отчитат повторни реакции) бяха разделени и филтрирани (т.е. с помощта на протокола RS_Subreads, минимална дължина на четене на полимераза = 50 bp, минимално качество на четене на полимераза = 75 и минимална дължина на subread = 50 bp), което е резултат от общо 107,5 Gb използваеми данни за последователността (общ брой на реакциите на полимераза = 11,6 M, брой на полимеразата N50 = 12,8 kb; общ брой на потапянията = 17,7 M, средна подкожна дължина = 5,7 kb, подпрочитане N50 = 8,0 kb). За MH63 бяха обработени данни от 317 SMRT клетки (174 Gb използваеми данни; 18,2 M полимераза чете, полимераза чете N50 = 12,1 kb; 26,8 M подпрочитания, средна дължина на подпрочитане = 5,5 kb, подпрочитане N50 = 7, 8 kb).
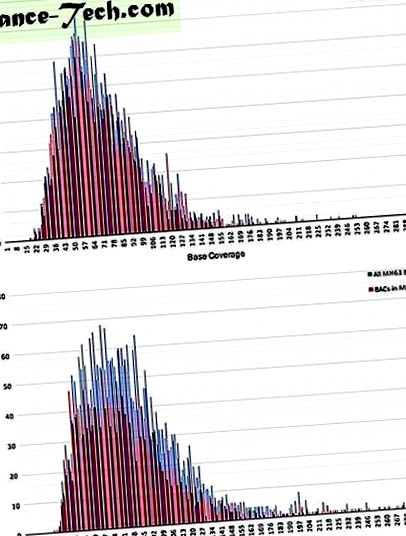
Съставяне на данни от PacBio и идентификация на BAC последователност
Изображение в пълен размер
Растителни материали, изграждане на ДНК библиотека за илюминирано секвениране
Използвахме също технологията за последователно четене на Illumin, за да секвенираме геномите ZS97 и MH63, използвайки подхода на целия геном (WGS). Растителните материали се отглеждат в оранжерия и се използват 4 седмични листа за извличане на геномна ДНК, като се използват стандартни процедури. Двойни библиотеки, включително малки вложки (
300 bp) и две библиотеки с големи вложки (5 kb, 10 kb) бяха подготвени с комплекти сдвоени краища и двойки Illumina (Таблица 1). Най-малко 5 μg геномна ДНК се фрагментира чрез пулверизиране със сгъстен азотен газ за кратки библиотеки за вмъкване на къса вложка. По-голямо количество висококачествена геномна ДНК (10-30 ug) се изисква, за да се изгради библиотека с дълга вложка на двойка двойка. Библиотеките с последователности на Illumina бяха подготвени съгласно протокола на производителя. Библиотеките бяха секвенирани на Illumina HiSeq 2000 съгласно стандартните протоколи на Ilulina (Illumina, Сан Диего, Калифорния). Общото количество данни за сурова последователност, генерирани за всеки сорт, беше
97, 5 Gb данни за ZS97 и
74.0 Gb данни за MH63. След поредица от стъпки за филтриране на данни, те бяха използвани за премахване на изкуствени показания, причинени от дублиране на PCR и замърсяване на адаптера, за общо 87.4 Gb висококачествени четения (> 200 ×) за ZS97 и
Получени са 67,9 Gb (> 170 ×) за MH63 (Таблица 1). Качеството на библиотеката беше проверено чрез определяне на разпределението на размерите на вложките и дълбочината на последователността. Действителните дължини на вмъкване бяха определени чрез картографиране на сдвоени краища към O. sativa subsp. японика cv. Референтен геном на Nipponbare (Nipponbare RefSeq) 17 .
Маса в пълен размер
Illumina чете предварителна обработка и de novo сглобяване
Използвахме нов хибриден подход, съчетаващ de novo сглобяване и референтни методи 18, за да съставим показанията на Illumina за всеки геном. Всички последователни показания от ZS97 и MH63 бяха коригирани с Medusa 19 и Quake 20. Коригираните четения бяха отсечени в краищата им, ако качеството на последователността им беше по-малко от 20 с помощта на fastx_tool_kit (//hannonlab.cshl.edu/fastx_toolkit/index.html) и броячите бяха премахнати с помощта на cutadpat 21 (//github.com/) Marcely/cutadapt /). След това обработените данни бяха картографирани в Nipponbare RefSeq с помощта на BWA 22. Всички показания, които са картографирани в непрекъснатата зона, са взети индивидуално и непрекъснатата зона на покритие е определена като блок. Дефиницията на блокове и суперблокове е същата, както е описана по-горе 23, с изключение на това, че нашата минимална дължина на суперблок беше 20 kb, а припокриването на суперблок беше 2 kb. Локално ние де ново компилирахме всички показания, събрани в суперблока, използвайки SOAPdenovo 24. Тествани бяха поредица от различни k-mers и останалите contigs с най-високите стойности на N50 бяха запазени. Получените контиги се сглобяват с AMOS 25, като се използват съответните им референтни хромозоми като водачи. Немапираните четения се картографират в генома на indica 9311 и се сглобяват по същата процедура.
След това Nucmer 27 беше използван за подравняване на всички суперконтроли към Nipponbare RefSeq. След това проверихме дълбочината на покритие и картографирането на пролуките между съседните зони. Картираните четения бяха избрани чрез удължаване на мостовите пропуски в геномите MH63 и ZS97 с 200 bp от двете страни на всяка празнина. Всички избрани числа в тези области са съставени с помощта на cap3 28. Сглобените контигове бяха подравнени с два непрекъснати суперконтрола и свързани на базата на тяхното последователно подреждане.
За да включим специфични последователности MH63 и ZS97, които не присъстваха в геномите Nipponbare и 9311, извършихме пълен сбор от ново геном, използвайки всички обработени показания на SOAPden 24 и след това подредихме де ново скелета, сглобени с комбинираните свръхпроводници и допълнително лигирахме съответните суперконтроли . И накрая, скелето беше извършено с помощта на SSPACE 29 и пропуските бяха запълнени с gapCloser (//sourceforge.net/projects/soapdenovo2/files/GapCloser/).
Окончателните статистически данни за комплектите WGS Illumina ZS97 и MH63 са показани в таблици 2 и 3 и са използвани за запълване на празнините между съседните PacBio контиги.
Маса в пълен размер
Маса в пълен размер
Изграждане на псевдомолекули в две стъпки
В първата стъпка всички данни за BAC последователност бяха вмъкнати в Master Puzzle Master 16 (GPM), за да се създадат базирани на PacBio последователности, като се използват физически карти и справочни ръководства за Nipponbare RefSeq 17. GPM е полуавтоматизиран тръбопровод, който е разработен за интегриране на данни за логическа връзка (напр. Физически карти) в последователности на скелета в последователности на хромозомния диапазон. В резултат на това 318 (ZS97) и 216 (MH63) сглобени контига бяха подредени и ориентирани, както и закрепени към съответните им хромозоми, след ръчна проверка, редактиране и отстраняване на излишъка. Финалните сглобки, базирани на PacBio, се състоят от последователности от 3862 (ZS97, включително 57 не-MTP) и 3254 (MH63, включително 77 не-MTP) уникални BAC.
Тъй като в това проучване използвахме стратегия за секвениране, базирана на физическа карта, пропуските в нашите WGP физически карти са основните причини за прекъсвания в базирани на PacBio възли. Следователно втората стъпка беше да се запълнят празнотите чрез интегриране на данните за сглобяване на WGS на Illumina. За да сведем до минимум въздействието на потенциално неверни данни в данните на Illumina, използвахме само contra Illumina, които биха могли да свържат напълно два съседни PacBio контакта в тази стъпка. Общо 81 празнини в ZS97 (8,988,328 bp) и 35 в MH63 (3,127,191 bp) бяха запълнени с 76 (ZS97) и 35 (MH63) Illumina contig последователности (Допълнителна таблица 4a-b). В резултат на това окончателните хибридни псевдомолекули (ZS97RS1 и MH63RS1) съдържаха 237 (включително 2 незатворени) и 181 (включително 2 непотвърдени) контига за ZS97 и MH63.
Записи на данни
Всички непоследователни данни са налични в iPlant Datastore (Таблица 4). Библиотеките на OSIZBa и OSIABa BAC или отделни BAC клонинги могат да бъдат получени от Ресурсния център на AGI за BAC/EST на адрес www.genome.arizona.edu/orders. Крайните последователности на BAC преди това са били съхранявани в GenBank под номера за присъединяване KG737749 - KG771717 (ZS97, цитиране на данни 1: GenBank KG737749 - KG771717) и KG702200 - KG737748 (MH63, цитиране на данни 2: GenBank KG702200 - KG737748).
Маса в пълен размер
Суровите данни за последователността на PacBio са достъпни в NCBI Short Read Archive (SRA) под номера за присъединяване SRP071597 (ZS97, Цитиране на данни 3: NCBI Sequence Read Archive SRP071597) и SRP071598 (MH63, Цитиране на данни 4: Архив за четене на последователност NCBI SRP071598) Всички данни за последователността на Illumina могат да бъдат намерени под номер за присъединяване SRP071944 (ZS97 и MH63, Цитиране на данни 5: NCBI Sequence Read Archive SRP071944). Поради неочаквана грешка на диска загубихме необработени данни за последователността на PacBio от 57 пула. За щастие, всички изпълнения на HGAP бяха архивирани в iPlant Datastore под „смъртни задачи“, където беше възможно да се получат филтрирани подслоеве на тези съответни групи. Събраните данни от Illumina са достъпни за събранието на NCBI под номера за присъединяване GCA_001618795 (ZS97, цитиране на данни 6: NCBI събрание GCA_001618795) и GCA_001618785 (MH63, цитиране на данни 7: събрание NCBI GCA_001618785).
Окончателните псевдомолекули на генома (версия 1) за всеки референтен геном се съхраняват в събранието на NCBI под номера за присъединяване GCA_001623345 (ZS97RS1, цитиране на данни 8: NCBI събрание GCA_001623345) и GCA_001623365 (MH63RS1, цитиране на данни 9: събрание NCBI_G0065).
Техническа проверка
По същество всяка геномна еквивалентна BAC библиотека е прясно отгледана в копирани набори от 384-ямкови плаки и е извършено триизмерно обединяване на бактериални клетки, последвано от клетъчен растеж и екстракция на плазмидна ДНК, използвайки алкална лизисна химия. ДНК комплектите се усвояват с рестрикционни ензими (EcoRI/MseI), последвано от лигиране на олигомери с размер на басейна, които са проектирани да локализират специфично адресите на BAC клонинги и да се свържат с последователностите. След амплифициране на смесените молекули беше извършено секвениране на Illumina и получените данни бяха анализирани, за да се идентифицира 50 bp последователен маркер за всеки специфичен адрес на BAC клонинга и да се генерират набори от ленти като вход към FPC. FPC се изпълняваше при високи строги настройки (HS): първо с 'толеранс = 0 [фиксиран], Cutoff = 1e-15', след това чрез избор на DQ (в 3 стъпки: Cutoff = 1e-18, 1e-21, 1e -24 ) се използва за разпространение на проблемни контигове. След създаването на получените HS PMs, извършихме стъпка за сливане Ends-to-Ends (Cutoff = 1e-9) и включихме етикетираните единични елементи в contigs (Cutoff = 1e-12), за да създадем карти с ограничена строгост (RS). Картите на WGP RS са ръчно модифицирани чрез интеграция с предишното ни покритие с ниски PMs 11 .
В стъпката GPM 'AssemblyRun' за създаване на BAC-базирани последователности на последователности, параметрите по подразбиране за обединяване на двете BAC последователности бяха 'minOverlapSeqToSeq = 1000 bp' и 'identitySeqToSeq = 99%', като се припокриват в края на двете последователности. Използвахме Nipponbare RefSeq 17 като референция, за да присвоим хромозомни номера на contig сглобяването, както и да ги подредим и ориентираме. В допълнение, само едно копие на излишната припокриваща се последователност беше запазено в сглобения contig, без да се дава предпочитание за определяне на коя част от BAC последователността беше запазена. Последователностите без пропуски обаче имаха по-висок приоритет от пропуските. Всички контигове бяха ръчно проверени и коригирани при необходимост с помощта на браузъра GPM 16. Когато използвахме събраните контигове на Illumina, за да запълним празнините между два базирани на BAC контига, избрахме само контиги на Illumin, които могат напълно да свържат два съседни BAC базирани контига и, което е важно, такива припокривания ('minOverlapSeqToSeq = 1000 bp' a 'identitySeqToSeq = 99% ") се изисква да се появи в края на двата контига." Когато в тези региони бяха открити излишни сайтове, последователностите на последователности, базирани на BAC, винаги се съхраняваха в крайни геномни възли.
Този документ е първото издание на първоначалните данни за сглобяването на оризови геноми ZS97 и MH63, а също така предоставя първите версии на два комплекта висококачествени псевдомолекули в научната общност. Технологиите за секвениране на ДНК и програмите за сглобяване на последователности се променят бързо и представените тук набори от данни съдържат няколко вида секвениране, които могат да се използват за разработване на нови методологии и софтуерни инструменти като тестови входни данни.
Повече информация
Как да цитирам тази статия: Zhang, J. et al. Генериране на два референтни генома оризов индика с данни за дълго четене PacBio и последователност на Illumina. Sci. Данни 3: 160076 doi: 10.1038/sdata.2016.76 (2016).
Цитиране на данни
GenBank KG737749 - KG771717 (2013)
GenBank KG702200 - KG737748 (2013)
Архив за четене NCBI SRP071597 (2016)
NCBI Sequence read archive SRP071598 (2016)
Архив за четене на NCBI Sequence SRP071944 (2016)
- Влюбен в Томаш Клус, той знаеше след две седмици, че иска бебе с Тамара
- Спиране на загубата на биологично разнообразие и създаване на здравословна и устойчива хранителна система в Европа
- Уелнес в хотел Fontana в Бърно с полупансион за двама и дете до 6 години безплатно, Чехия - Бърно
- Yelp използва машинно образование, за да създаде списък с любими храни
- Може да има четири от двете убийства